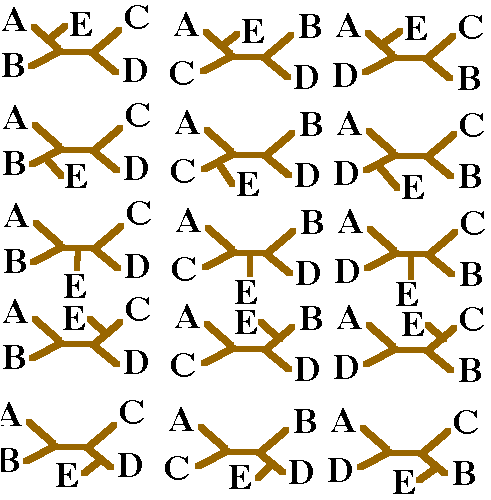Princípios gerais do método de parcimônia
O princípio de parcimônia é um princípio
filosófico largamente empregado na Ciência. Foi proposto por
um filósofo inglês, Ockam no século XVII e seu enunciado
é aproximadamente o seguinte: Se existe mais de uma explicação
para uma dada observação, devemos adotar aquela mais simples.
O método de máxima parcimônia
foi empregado em análises de seqüências moleculares com
o propósito de reconstrução de árvores filogenéticas
como uma simplificação do método de máxima
verossimilhança por Eck e Dayhoff (1967). Já havia sido empregado
implicitamente com o propósito de explicar a origem das inversões
aninhadas nos cromossomos politênicos de espécies de Drosophila,
em 1936 por Sturtervant e Dobzhansky.
1. O princípio da parcimônia nas reconstruções
filogenéticas a partir de dados de seqüências macromoleculares.
A aplicação do princípio de máxima
parcimônia nas reconstruções filogenéticas é
conceitualmente muito simples: Devemos escolher, dentre as árvores
possíveis, aquela com o menor número de substituições
que seja compatível com as seqüências observadas. Entretanto,
a implementação é complexa, pois o número de
árvores possíveis pode atingir valores muito altos.
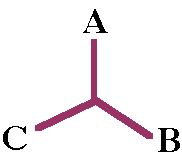
Para 3 seqüências, existe apenas uma
árvore possível (que sejam bifurcantes, árvores tais
como A--B--C não são consideradas):
Para 4 seqüências, existem 3 árvores
(bifurcantes) possíveis.
 Para 5 seqüências existem 15 árvores
(bifurcantes):
Para 5 seqüências existem 15 árvores
(bifurcantes):
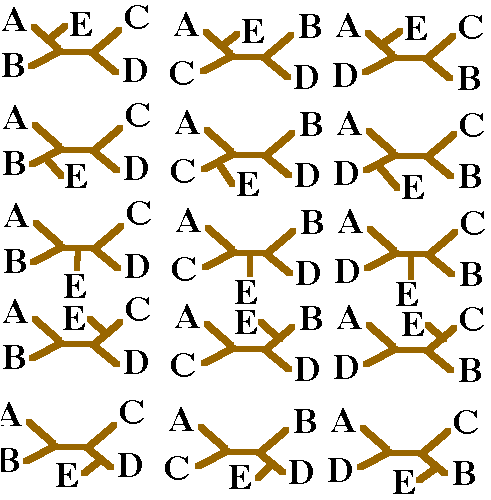 O número de árvores cresce muito rapidamente
à medida que aumenta o número de seqüências. Você
deve ter notado que para saber o número de ávores com N seqüências,
multiplica-se o número de árvores com N-1 seqüências
pelo número de ramos presentes em uma árvore com N-1 seqüências.
Para 10 seqüências,mais de um milhão de árvores
são possíveis. Para se atingir o número de partículas
elementares do Universo conhecido (~1080), bastam 59 seqüências!
E isso contando apenas as árvores sem raiz e estritamente bifurcantes.
O número de árvores cresce muito rapidamente
à medida que aumenta o número de seqüências. Você
deve ter notado que para saber o número de ávores com N seqüências,
multiplica-se o número de árvores com N-1 seqüências
pelo número de ramos presentes em uma árvore com N-1 seqüências.
Para 10 seqüências,mais de um milhão de árvores
são possíveis. Para se atingir o número de partículas
elementares do Universo conhecido (~1080), bastam 59 seqüências!
E isso contando apenas as árvores sem raiz e estritamente bifurcantes.
2. Algoritmos para a reconstrução filogenética com
máxima parcimônia.
O algoritmo mais simples de se entender mas que também
é o de maior exigência computacional é o de busca
exaustiva, onde todas as árvores possíveis são
verificadas. Na prática o emprego desse algoritmo é inviável
para muitas seqüências. Existe também um algoritmo exato
, "branch and bound" (que assegura que a árvore mais parcimoniosa
é encontrada), que verifica apenas um subconjunto delas. Existem
ainda os algoritmos heurísticos, que, embora não consigam,
com certeza, determinar a árvore mais parcimoniosa, analisam um
subconjunto muito reduzido das árvores possíveis mas que
são mais prováveis de conter a mais parcimoniosa, de
acordo com critérios variáveis conforme o algoritmo. Entre
este temos o "branch-swaping", o "star decomposition"
e o "nearest neighbor interchange".
3. Variações do método de máxima parcimônia.
O método de parcimônia, em sua forma mais
simples, tem como sua principal vantagem, segundo seus defensores,
a independência de modelos de evolução. Seus detratores
argumentam, no entanto, que na verdade existe um modelo implícito,
por exemplo, no qual todos os sítios de uma seqüência
macromolecular tiem pesos iguais, de que não é considerada
a diferença empírica entre taxas diferentes de transições
e transversões, etc, em uma analogia com o modelo de Jukes e Cantor
(1969).Certas variantes do método de parcimônia foram propostas
para admitir modelos explícitos de evolução. Temos
abaixo alguns exemplos:
a) Parcimônia de transversão.
Nesse método, as mudanças que envolvem
transições são simplesmente ignoradas da análise
b) Emprego de uma matriz de passos
Para contemplar, por exemplo, diferenças
entre transições de transversões, são empregadas,
a
priori, uma contagem diferente para transições e transversões.
Por exemplo, com o emprego de uma matriz desse tipo:
|
DE
|
|
PARA
|
|
A
|
T
|
C
|
G
|
|
A
|
-
|
2
|
2
|
1
|
|
T
|
2
|
-
|
1
|
2
|
|
C
|
2
|
1
|
-
|
2
|
|
G
|
1
|
2
|
2
|
-
|
As transições contam um passo e as transverções
contam 2 passos. Uma matriz para seqüências de aminoácidos
em proteínas teria a dimensão 20,20.
Outros detalhes poderão ser encontrados em Swofford et al. (1996)
e Miyaki et al. (2000).
Página principal